Wirkstoff-Suche auf neuem Level
Nach dem erstmaligen Nachweis, dass sich aus uraltem Zahnstein bakterielle Wirkstoffe herstellen lassen, will das Projekt Paläobiotechnologie seinen Forschungs-Output vervielfachen. Dazu haben die Forschenden Methoden und Plattformen entwickelt, um komplexe Arbeitsschritte zu automatisieren.
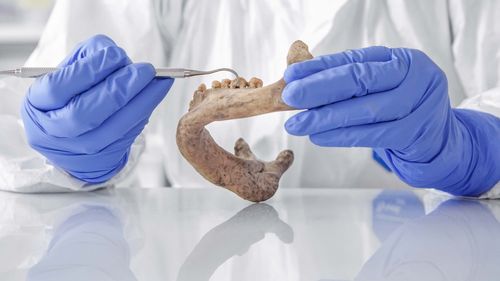
Das Projekt Paläobiotechnologie in Jena macht rasante Fortschritte. Vor zwei Jahren wiesen die Forschenden um Chemiker Pierre Stallforth und Archäogenetikerin Christina Warinner nach, dass ihre Idee funktioniert: In einer viel beachteten Studie im renommierten Fachmagazin «Science» zeigten sie erstmals auf, dass es möglich ist, aus dem Zahnstein von Frühmenschen Naturstoffe wieder herzustellen, die vor 100 000 Jahren von Bakterien produziert worden waren.
Mit eigens dafür entwickelten Methoden spürten sie Erbgutfragmente auf, setzten sie zusammen und rekonstruierten so die Genome zahlreicher Bakterienarten. Daraus wiederum rekonstruierten sie einen Bauplan für Enzyme, die Naturstoffe produzieren. Sie bauten diese Gene in lebende Bakterien ein – und tatsächlich bildeten diese bisher unbekannte Wirkstoffe.
«Wenn Leute hören, dass wir mit altem Erbgut arbeiten, denken sie oft, dass wir nicht genug davon finden», sagt Christina Warinner. «Aber das ist nicht wahr. Im Gegenteil: Wir haben enorm viele Daten – so viele, dass wir niemals alle testen können.» Um so viele wie möglich untersuchen und die besten Kandidaten finden zu können, ist es entscheidend, die komplexen Arbeitsschritte bei der Suche und der Herstellung solcher prähistorischer Stoffe zu automatisieren.

Drei Automatisierungsschritte
Genau darauf haben die Forschenden im vergangenen Jahr ihren Fokus gelegt. Grob gesagt, handelt es sich um drei Automatisierungsschritte: Der erste ist es, das Erbgut aus den Proben zu extrahieren und zu sequenzieren. «Die meisten dieser Arbeiten werden bereits von Robotern durchgeführt», erzählt Warinner. «Das hat unseren Durchsatz erhöht und zu einer Vereinheitlichung geführt.»
Im zweiten Schritt, ergänzt Pierre Stallforth, gehe es darum, möglichst viel aus diesen Daten herauszuholen. Die Forschenden haben diverse Software-Tools und Pipelines zur Automatisierung der Datenanalyse entwickelt. Diese Programme identifizieren beispielsweise DNA-Sequenzen, welche die Informationen zur Herstellung antimikrobieller Stoffe kodieren könnten.
Als dritten Schritt schliesslich hat das Leibniz-Institut für Naturstoff-Forschung und Infektionsbiologie in Jena eine weltweit einzigartige Automatisierungsplattform aufgebaut, um aus den neu gefundenen Sequenzen Moleküle zu erstellen und diese standardisiert und mit hohem Durchsatz zu testen – zum Beispiel bezüglich ihrer antibiotischen oder antimykotischen Wirkung.
«Wir haben verschiedene Fachgebiete – vom Programmierer bis zur Chemikerin – zusammengebracht, um die bestmöglichen Tools zu entwickeln», sagt Warinner. Was sind die wichtigsten Merkmale von Naturstoff-Kandidaten? Wie lassen sich systematische Wege finden, um vielversprechende Sequenzen herauszufiltern? Welche biologischen Merkmale sind besonders interessant? Wie stimmt man die verschiedenen Automatisierungsschritte aufeinander ab?
«Solche Fragen zu beantworten und solche Tools aufzubauen, braucht viel Zeit», sagt Pierre Stallforth. «Aber wenn es einmal reibungslos funktioniert, werden wir in der Lage sein, mithilfe einer verlässlichen Plattform enorm viel aus unseren Daten herauszuholen.» Dafür sei die langfristige Unterstützung der Werner Siemens-Stiftung Gold wert. «Viele andere Förderinstrumente laufen bloss über zwei oder drei Jahre – in dieser Zeit ist es unmöglich, solche grundlegenden Plattformen zu entwickeln.»
Zahnsteinproben aus aller Welt
Daneben ging die Arbeit mit archäologischen Zahnstein-Proben weiter. So haben die Forschenden Daten einer ganzen Reihe neuer Neandertaler-Skelette aufgenommen und ihre archäologische Erbgut-Sammlung deutlich ausgeweitet. «Anfangs stammten die meisten unserer Proben aus Europa», sagt Christina Warinner. «Jetzt haben wir beispielsweise neue Projekte in Asien und eine grosse Studie in Ozeanien.» Das Fernziel ist es, Daten von so vielen Individuen zu bekommen, dass die Untersuchung globaler Muster in der frühzeitlichen Zahnstein-Biodiversität möglich ist.
Auch personell gibt es gute Neuigkeiten: Christina Warinner wurde an der renommierten Harvard University zur ordentlichen Professorin mit eigenem Lehrstuhl befördert. Damit haben nun sowohl sie als auch Pierre Stallforth eine akademische Festanstellung. «Das ist grossartig», sagt Warinner. «Nun haben wir beide eine solide Grundlage, auf der wir unsere Arbeit in der Paläobiotechnologie weiter aufbauen und ausbauen können.»